自從SARS-CoV-2於全球擴散感染,於2020年底全球感染人數約達一億例時,許多重要突變位點逐漸顯現,其中某些特定變異位點使病毒增加傳染力,或使病毒逃脫特定中和抗體之辨識。
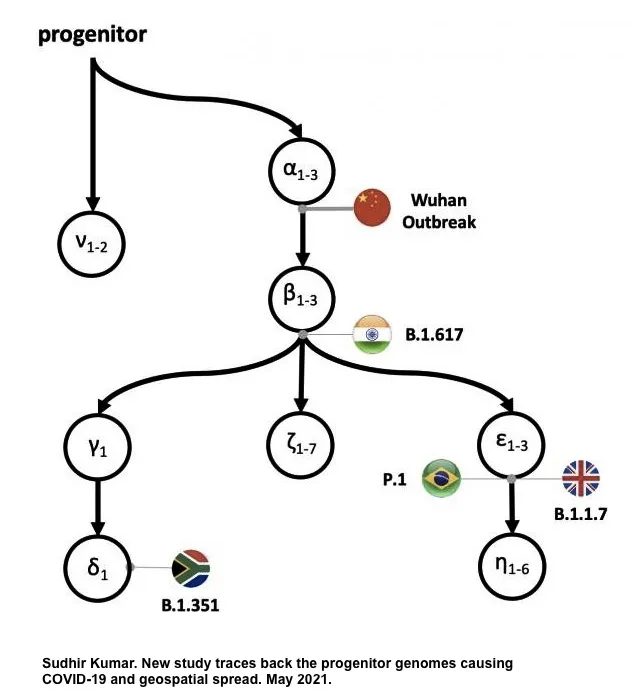
SARS-CoV-2基因研究的濫觴始於中國與澳洲團隊,合作發表SARS-CoV-2基因體分析 [1],緊接著世界各地的科學家紛紛投入病毒相關之研究,並且也積極發展檢測試劑與疫苗開發。而科學家關注的問題之一,便是了解SARS-CoV-2突變之機轉。與大部分RNA病毒不同的是,SARS-CoV-2和SARS-CoV、MERS病毒一樣,擁有校對能力之聚合酶 (proof-reading polymerase) - Nsp14 [2],因此病毒突變能力較為溫和,非快速突變之病毒。然而,當SARS-CoV-2在全球肆虐,不斷在群眾間感染與複製,如此便給病毒相當多突變機會。基因定序結果顯示,在病毒刺突蛋白 (spike protein)基因中出現數個重要的突變,這些突變之所以被重視,原因為刺突蛋白是負責與細胞接合、幫助病毒進入細胞之蛋白,也與病毒之致病機轉有關。在刺突蛋白眾多變異中,發現D614G (Asp614Gly)此變異被保留下來,逐漸取代舊有的D614,但此突變似乎只影響病毒感染力,使其複製能力上升,並不改變病毒毒力 [3],因此學界對此突變的關注,從開始研究後,遂隨時間逐漸消退;直至2020年底,當全球感染人數即將突破一億例時,科學家於一個月內,分別偵測到三株令人憂心之突變病毒株:B.1.1.7 (英國變種) [4]、B.1.351 (501Y.V2) (南非變種) [5] 與 B.1.1.24/P.1 (巴西變種) [6],並且這三種變異株在刺突蛋白上表現一系列全新組合的變異位點,並且皆帶有D614G突變,同時也發現,這三株變異株出現的地區,其感染病例數也大幅增加 [7],因此馬上引起流病專家注意,也預測此三種變異病毒株,將讓原本群體免疫低下的社會,帶來新一波的疫情。
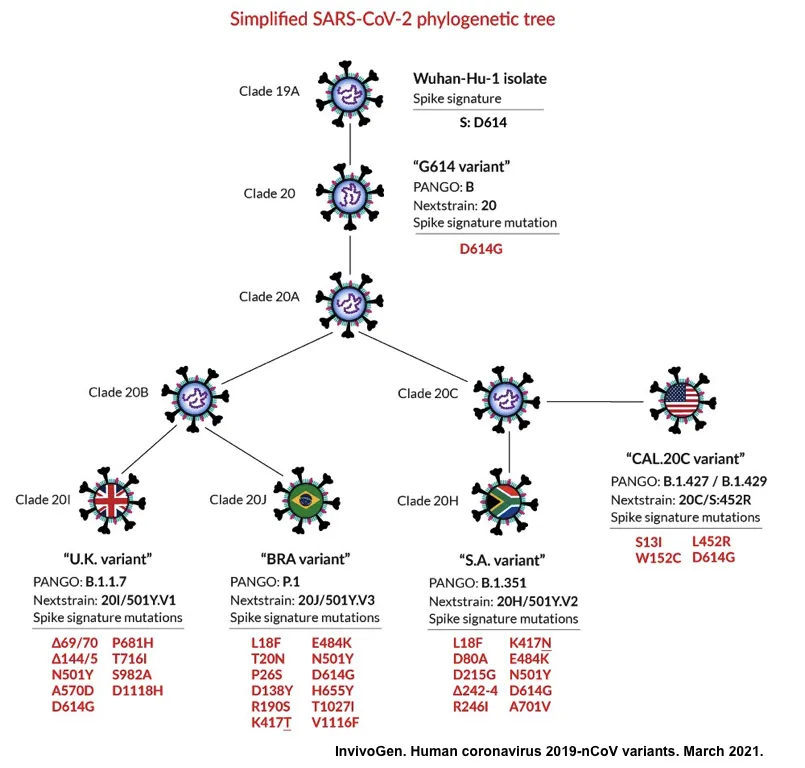
令專家憂慮的是,在疫情爆發初期,許多疫苗研發便是針對原始分離株 Wuhan-Hu-1之刺突蛋白進行開發,而初步實驗結果顯示,Wuhan-Hu-1刺突蛋白誘發之中和抗體似乎無法有效中和變異株,在南非的疫苗臨床試驗也顯示疫苗保護力不如預期,顯示疫苗株與當地流行株,在免疫辨識上已出現差異 [8, 9]。也正因如此,科學家更密切關注SARS-CoV-2刺突蛋白基因的演化,在眾多突變中,位於刺突蛋白受體結合區 (receptor binding domain, RBD)之變異特別受到重視,此區域負責病毒結合細胞受體ACE2,以利病毒進行感染,因此宿主細胞主要也透過此區域辨識病毒抗原,受體結合區為重要之免疫抗原區,也是中和抗體結合的所在。而在這些區域引起注意之突變位點包含K417N、E484K與N501Y [10];這三個突變皆不斷在流行株中出現,明顯有演化優勢,病毒不但傾向轉變為這些位點,帶有突變之病毒也較容易與ACE2結合,造成病患體內病毒量較高 [11, 12, 13]。以E484K為例,此突變讓病患感染病毒後更不易痊癒,也使病毒更容易逃脫中和抗體的辨識 [14, 15]。病毒也傾向將刺突蛋白之N端進行突變造成部分序列遺失;英國變異株B.1.1.7之刺突蛋白N端H69/V70兩個胺基酸的遺失,造成病毒感染力上升 [16]。除了上述三種變異病毒株外,美國加州所分離的B.1.427/B.1.429,其帶有位於刺突蛋白受體結合區的L452R,此突變被認為能增加病毒之傳染力,且降低抗體之中和效力 [17]。而近期讓全球聞之色變的印度變異株B.1.617,在其大約15個突變位點中,帶有L452R及E484Q兩個重點突變,目前科學家認為B.1.617會降低抗體中和能力,導致疫苗保護力下降 [18]。而除了胺基酸變異外,病毒之基因片段重組,也是病毒演化模式之一;不同來源之SARS-CoV-2在感染同一宿主後,可能於宿主體內進行重組,而演化為致病力更強之變異株 [19],這也是目前學界專家最憂心的議題。
現階段疫苗對大多數人仍有保護力,病毒尚未逃脫免疫辨識導致的抗原移型 (antigenic shift),因此疫苗所誘發之抗體,仍可使接種者產生中和抗體或減少重症發生 [20],但要達到群體免疫所需要施打的人口比例,目前仍屬推測,尚未有實驗驗證;隨著病毒不斷突變,若無法在疫苗有效力之前,達到群體免疫,徹底切斷病毒感染鍊,一旦病毒在未帶有抗體之人群間傳染,不斷複製且加速病毒抗原變異,將使疾病控制清況變的更加嚴峻 [21]。
所幸在全球各界學者相互合作之下,SARS-CoV-2病毒基因分析、特殊變異位點與不同物種間序列差異都已有詳盡研究,提供疫情監測與防治重要資訊 [22]。進一步則需探討這些突變是否影響病毒毒力或傳染力,藉由深度突變掃描技術 (deep mutational scanning)與結構模擬,可推測突變對於病毒致病機轉之影響 [23, 24],但仍然缺乏全面性的評估,對於真實情況仍難以掌握。要探知疫苗保護效力,最可信的方式為偵測其中和抗體效價,但目前對於這類資訊尚且不足,疫苗相關之大規模血清研究仍以快篩為主,因此常有失專一性與敏感度 [25];為此,我們需要更好的標準化工具來評估抗體中和效力,以檢視疫苗是否對於突變病毒有所保護能力 [26]。
而SARS-CoV-2是七種人類冠狀病毒之一,在SARS-CoV-2出現之前,其中四種冠狀病毒已在人類社群間傳播多年,造成輕微疾病,因此一直未被世人重視。儘管如此,人類冠狀病毒依然不斷演化 [27, 28],為逃脫人體免疫系統,不斷突變修正表面抗原,特別是受體結合區。如同SARS-CoV-2,人類冠狀病毒之受體結合區亦是免疫系統辨識的高度變異區。因此設計SARS-CoV-2疫苗設時,常參考過去人類冠狀病毒演化的研究,以這些區域為目標;但也因為這些區域具高度變異性,因此序列相對不保守,一旦出現逃脫的變異株,便可使疫苗失效。為了克服這個困難,科學家設計兩種以上的抗原製成疫苗,以追加第二劑注射的方式,希望能讓接種疫苗者產生多樣性的抗體 [29]。另一方面,研究學者也努力朝向廣效性疫苗的開發進行研究,希望找到病毒高度保留區並且具有良好免疫原性的片段,能誘發廣效性抗體,用以作為疫苗標的區域 [30]。為此,許多新策略被學界思考與應用,例如嵌合性刺突蛋白,此蛋白表達多種變異株受體結合區之重要變異位點,並且保留多個免疫抗原位,因此有潛力發展成單一劑量之廣效性疫苗。然而如何正確設計出良好的嵌合性蛋白,並且能誘發廣效性且具中和能力的抗體,仍需要足夠資訊,對於抗體、抗原決定位點與突變位點都需要全方面的了解,同時也需要針對這些抗原位置與之對應的抗體,進行高通量中和試驗,以篩選合適之突變位點。
為了加速研究進行,The Native Antigen Company 提供中和試驗研發套組,包含四種關鍵蛋白:SARS-CoV-2刺突蛋白之受體結合區、帶標記ACE2、正控制與負控制抗體,不但操作容易、快速且適用於96孔盤。
更多資訊:https://thenativeantigencompany.com/products/sars-cov-2-neutralization-assay-development-kit-rbd-ace2/
The Native Antigen Company 同時也提供多種突變位點且具醣基化、正確構型之刺突蛋白,提供各種抗體辨識或進行抗體結合試驗;除此之外,也供應多種SARS-CoV-2相關試劑進行多樣測試。
更多資訊:https://thenativeantigencompany.com/coronavirus-reagents/
References:
- https://www.who.int/china/news/detail/09-01-2020-who-statement-regarding-cluster-of-pneumonia-cases-in-wuhan-china
- Fran Robson, et al., Coronavirus RNA proofreading: molecular basis and therapeutic targeting. Mol Cell. 2020.
- Erik Volz, et al., Evaluating the effects of SARS-CoV-2 spike mutation D614G on transmissibility and pathogenicity. Cell. 2021.
- https://virological.org/t/genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-manaus-preliminary-findings/586
- https://virological.org/t/preliminary-genomic-characterisation-of-an-emergent-sars-cov-2-lineage-in-the-uk-defined-by-a-novel-set-of-spike-mutations/563
- Houriiyah Tegally, et al., Emergence and rapid spread of a new severe acute respiratory syndrome-related coronavirus 2 (SARS-CoV-2) lineage with multiple spike mutations in South Africa. medRxiv. 2020.
- PrernaPriyaa and Asheesh Shanker. Coevolutionary forces shaping the fitness of SARS-CoV-2 spike glycoprotein against human receptor ACE2. Infect Genet Evol. 2021.
- https://ir.novavax.com/news-releases/news-release-details/novavax-covid-19-vaccine-demonstrates-893-efficacy-uk-phase-3
- https://www.jnj.com/johnson-johnson-covid-19-vaccine-authorized-by-u-s-fda-for-emergency-usefirst-single-shot-vaccine-in-fight-against-global-pandemic
- Zijun Wang, et al., mRNA vaccine-elicited antibodies to SARS-CoV-2 and circulating variants. Nature. 2021.
- Nicholas G. Davies, et al., Increased mortality in community-tested cases of SARS-CoV-2 lineage B.1.1.7. Nature. 2021.
- Robert Challen, et al., Risk of mortality in patients infected with SARS-CoV-2 variant of concern 202012/1: matched cohort study. The BMJ. 2021.
- Renato Mendes Coutinho, et al., Model-based estimation of transmissibility and reinfection of SARS-CoV-2 P.1 variant. medRxiv. 2021.
- Pengfei Wang, et al., Antibody resistance of SARS-CoV-2 variants B.1.351 and B.1.1.7. medRxiv. 2021.
- Dami A. Collier, et al., Sensitivity of SARS-CoV-2 B.1.1.7 to mRNA vaccine-elicited antibodies. Nature. 2021.
- Kevin A. Brown, et al., S-gene target failure as a marker of variant B.1.1.7 among SARS-CoV-2 isolates in the greater Toronto area, December 2020 to March. JAMA. 2021.
- Veronika Tchesnokova, et al., Acquisition of the L452R mutation in the ACE2-binding interface of Spike protein triggers recent massive expansion of SARS-Cov-2 variants. bioRxiv. 2021.
- CDC. SARS-CoV-2 variant classifications and definitions. 2021.
- Anastasia Ignatieva. et al., Evidence of ongoing recombination in SARS-CoV-2 through genealogical reconstruction. medRxiv. 2021.
- Zhuoming Liu, et al., Landscape analysis of escape variants identifies SARS-CoV-2 spike mutations that attenuate monoclonal and serum antibody neutralization. medRxiv. 202
- Chloe Rees-Spear, et al., The effect of spike mutations on SARS-CoV-2 neutralization. Cell Rep. 2021.
- Andrew L. Valesano, et al., Temporal dynamics of SARS-CoV-2 mutation accumulation within and across infected hosts. bioRxiv. 2021.
- Tyler N. Starr, et al., Deep mutational scanning of SARS-CoV-2 receptor binding domain reveals constraints on folding and ACE2 binding. Cell. 2020.
- Allison J. Greaney, et al., Complete mapping of mutations to the SARS-CoV-2 spike receptor-binding domain that escape antibody recognition. Cell Host and Microbe. 2021.
- Zhong Yao, et al., A homogeneous split-luciferase assay for rapid and sensitive detection of anti-SARS CoV-2 antibodies. Nat Commun. 2021.
- John R. Mascola, et al., SARS-CoV-2 viral variants—tackling a moving target. JAMA. 2021.
- A. Callow, et al., The time course of the immune response to experimental coronavirus infection of man. Epidemiol Infect. 1990.
- W. Schmidt, et al., Rises in titers of antibody to human coronaviruses OC43 and 229E in Seattle families during 1975-1979. Am J Epidemiol. 1986.
- Qian He, et al., Heterologous prime-boost: breaking the protective immune response bottleneck of COVID-19 vaccine candidates. Emerg microbes and infect. 2021.
- Lianpan Dai, et al., A universal design of betacoronavirus vaccines against COVID-19, MERS, and SARS. Cell. 2020.
